聴講希望の方は、下記Zoom参加登録用アドレスからご登録ください。
https://us02web.zoom.us/meeting/register/tZMpcu-srz8qH9SfD-NtCBWzHythcHbAWqb0
要旨
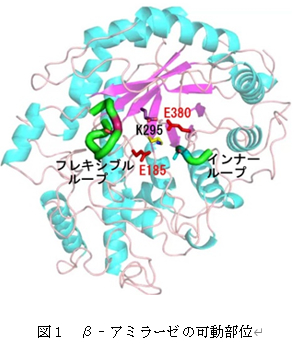
現在、多くの酵素の立体構造がX線結晶構造解析によって明らかにされ、その機能が検討されている。しかし、結晶構造には様々なアーチファクトが含まれている。第一に、目的タンパク質の結晶内でのパッキングによっては、正常な基質複合体の形成が困難なことがある。第二に、凍結結晶を用いた構造解析では凍結と凍結保護剤の影響が無視できない場合が多い。一般に凍結により、結晶格子の収縮と結晶のモザイク性の増大が生じる。その結果、リガンドとの結合に変化が生じることがある。さらに、pH変化を追跡する場合、結晶格子の収縮も含めて凍結結晶のpHを再現することが難しい。凍結の関係する問題についてはキャピラリー測定等の非凍結測定法を用いると解決できる。発表者らはダイズβ-アミラーゼの非凍結晶を用いて基質アナログ複合体のpH変化の解析を行って、本酵素の反応機構について検討した。β-アミラーゼはデンプンの非還元末端からβ-アノマーのマルトースを遊離する酵素で、ダイズ酵素の触媒残基は酸触媒のGlu186 と塩基触媒のGlu380 である。活性部位にはフレキシブルループ(96-104)とインナーループ(341-344)および可動な側鎖を持つLys295が存在する(図1)。これらの可動部位の役割を明らかにするために、G1(グルコース)、G2(マルトース)およびG5(マルトペンタオース)を用いて、pHと基質アナログ濃度を変化させて構造解析を行った。G1はpH3~4では、サブサイト-2と+2の2個所に結合するが、pH5以上ではサブサイト-2, +1, +2の3個所に結合する。サブサイト+1へのG1の結合はインナーループのアポ型から複合体型への構造変化と同期し、そのpH依存性から塩基触媒であるGlu380(pKa = 3.6)の関与が示唆された。G2はサブサイト-2~-1と+1~+2および+2~+3に連続して結合し、-1サイトのGlc残基はpH3~4では、α/βのアノマーの4C1のイス型の構造を取るのに対して、pH5以上ではαアノマーの1,4Bのボート型の構造を取る。イス型からボート型への構造変化のpH依存性からGlu380の解離が関係すると推定された。次にE380A変異体を用いて基質であるG5との結合のpH依存性を調べた。G5はサブサイト-2~+3に結合するが、pHによってサブサイト-1のGlc残基の結合位置が変化し、その変化には酸触媒であるGlu186(pKa = 8.1)が関与すると推定された。以上の結果から、触媒残基の2つのGlu残基の解離状態が酵素と基質の構造変化に大きく関わり、触媒反応の前後でサブサイトの親和力を変化させて酵素反応を有利に進めることが明らかになった。
印刷用PDFファイル(200,694 バイト) | ページ先頭へもどる
ページ先頭へもどる
2023年9月15日作成/10月12日更新


